DEGRADAÇÃO PROTEICA: PROTEASSOMOS
Carlos Augusto B.M. Normann
(Original publicado em NORMANN, CABM (ORG.) Práticas em biologia celular. 2. ed. Porto Alegre: Sulina; Porto Alegre: Editora Universitária Metodista IPA, 2017. 303 p.)
INTRODUÇÃO
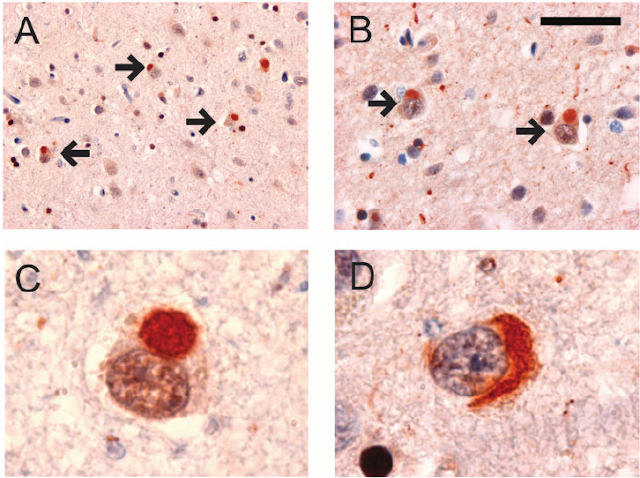 |
| Inclusões citoplasmáticas neuronais imunorreativas à ubiquitina nas camadas profundas do neocórtex cerebral (A e B). As inclusões variaram em forma e incluíram formas redondas (C), ovais e crescênticas (D). Imuno-histoquímica de ubiquitina. Barra de escala; A, 75 m; B, 38 m; C e D, 8 m m. Original? McKenzie et al, 2008. Atypical frontotemporal lobar degeneration with ubiquitin-positive, TDP-43-negative neuronal inclusions. |
A degradação
proteica pode ocorrer nos lisossomas, endossomas secundários, no
retículo rugoso e no citoplasma. Pode ocorrer pela ação de proteinases das mitocôndrias, das membranas, pode estar ligadas às enzimas denominadas caspases, ou depender do cálcio, como é o caso das calpaínas, que degradam moléculas do citoesqueleto, ajudando em seu reciclo. A célula apresenta, pois, mais de uma
forma de reciclar proteínas. Uma delas envolve o marcar as proteínas para degradação, dirigindo-as a um
complexo enzimático de desmanche proteico. Os proteassomas são
esses complexos enzimáticos citoplasmáticos, compostos por diversas
classes de proteases dispostas em torno de um conduto central. As
proteínas citossólicas, quando danificadas, mal dobradas,
desestruturadas ou com aminoácidos oxidados, são degradadas no
cilindro do proteassoma, onde estão os sítios ativos das proteases,
gerando oligopeptídios de vida curta.
 |
| Visão resumida dos sistemas intracelulares de proteólise. |
As proteínas
a serem degradadas são endereçadas aos proteassomas pela marcação
com a ubiquitina. A
ubiquitinação
é um processo de modificação pós-traducional de proteínas.
Composta por 76 aminoácidos, a ubiquitina é uma proteína que pode
ser conjugada às proteínas-alvo de forma única ou em cadeias de
poliubiquitinas (conjugação adicional de ubiquitinas) e essa
marcação pode sinalizar para mecanismos proteolíticos ou não
proteolíticos. Os primeiros incluem a degradação proteassômica
para a eliminação de certos substratos visando à progressão
adequada do ciclo celular, regulação transcricional, controle de
qualidade das proteínas, correta transdução de sinais e ritmos
circadianos. Dentre os mecanismos não proteolíticos se encontram a
endocitose de proteínas, tráfego intracelular, regulação da
transcrição mediada por cromatina, reparo do DNA e interligação
dos complexos de sinalização. O processo de ubiquitinação ocorre
basicamente em três etapas: 1), A enzima ativadora de ubiquitina
(E1) possui um resíduo cisteína que é ligado ao resíduo
C-terminal da ubiquitina, formando uma ligação tioéster. Essa
reação necessita de adenilação de um resíduo glicina da
ubiquitina pela ligação de um AMP proveniente da hidrólise do ATP,
liberando pirofosfato. 2), Uma vez conjugada à E1, ocorre a
transferência da molécula de ubiquitina para uma cisteína da
enzima conjugadora (E2), formando outra ligação tioéster; e, 3),
finalmente, a enzima ubiquitina-ligase (E3) é capaz de se ligar
tanto na E2 como à proteína alvo de maneira a catalisar a ligação
da porção C-terminal da molécula de ubiquitina na porção amina
de uma lisina presente na proteína alvo. Um dos sinais
para a ubiquitinação é a presença de resíduos desestabilizadores
na porção N-terminal, mas outros sítios para ubiquitinação
existem também na porção C-terminal. Depois de marcadas, as
proteínas entram nos proteassomas pela abertura do cilindro, que é
parcialmente tampada por um complexo proteico que parece funcionar
como um filtro seletivo das proteínas que devem entrar no conduto
central do cilindro.
O processo
de degradação proteica, no interior do proteassoma, consome
energia, oriunda da hidrólise do ATP. Depois de degradada a
proteína, as ubiquitinas e o proteassoma são liberados e podem ser
reutilizados. Existem muitas cópias de proteassomas nas células. O
proteassoma permite que a concentração de diferentes proteínas
seja modificada rapidamente, de acordo com as demandas funcionais da
célula. Assim, certas proteínas têm um curto período de
meia-vida, enquanto outras têm sua meia-vida prolongada, por
possuírem aminoácidos especiais na sua porção N-terminal que
impedem a ubiquitinação.
Um exemplo
está na transição da metáfase para a anáfase; a degradação de
coesinas (proteínas que mantêm as cromátides-irmãs juntas) é
fundamental para a distribuição das cromátides entre as
células-filhas e a manutenção da ploidia correta. No entanto, a
ubiquitinação de proteínas também serve para modificá-las, ao
invés de ativar sua degradação; como exemplo temos a ubiquitinação
da histona H2A. Embora relativamente recentes, estudos mostram que a
degradação pelo eixo ubiquitina-proteassomo parece afetar
praticamente todos os processos celulares.
A
sinalização por ubiquitina e suas cadeias tem um papel
não-proteolítico no transporte via membrana, na estrutura e
transcrição da cromatina, no reparo do DNA e em diversas outras
vias sinalizadoras. Como vimos, ao contrário de outras vias de
degradação proteica, a ubiquitinação envolve consumo de energia.
A degradação proteica é feita em uma série de etapas que resultam
na ubiquitinação da proteína a ser destruída; esse processo
permite que a célula elimine proteínas de modo bastante específico,
e é essa regulação que exige hidrólise de ATP.
Tanto a
ubiquitina como sua via proteolítica participam de diversos outros
processos celulares; a ubiquitinação da ciclina em G1 mitótico
ajuda a regular o ciclo celular; é responsável por estimular a
diferenciação dos linfócitos T e B. A ubiquitina desempenha papéis
na biogênese ribossomal, na modulação de receptores celulares, na
expressão gênica, no reparo do DNA, em resposta ao estresse, na
neurogênese, na memória de longo prazo, nos ritmos circadianos e em
determinadas patologias, como o mal de Alzheimer. A ubiquitina
participa da mitose, dando direcionalidade a ela, por meio do APC
(Anaphase-Promoting Complex), uma enzima E3 que provoca indiretamente
a degradação da coesina, um complexo que mantém as
cromátides-irmãs unidas, permitindo assim a separação delas. A
via proteolítica da ubiquitina-proteassoma difere das demais vias
proteolíticas devido a seu consumo de ATP.
Há cinco
alvos principais da ubiquitina:
1. Receptores da membrana
plasmática;
2. Moduladores de
crescimento e supressores de tumor;
3. Ativadores e inibidores
da transcrição;
4. Reguladores do ciclo
celular;
5. Proteínas
mutantes ou danificadas.
Trinta por
cento das proteínas recém-sintetizadas de uma célula são
quebradas via proteassomas por não terem sido aprovadas pelo
rigoroso controle de qualidade celular. Sabe-se que as chaperonas
atuam no reconhecimento de proteínas mal dobradas. Essas proteínas
auxiliam no correto dobramento de outras proteínas nascentes;
diversas chaperonas necessitam de ATP, e algumas proteínas são
dependentes de chaperonas para atingirem a conformação adequada. Há
evidências de que as chaperonas participem ativamente do processo de
ubiquitinação, interagindo com as enzimas E3, que ligam a
ubiquitina à proteína-alvo, sugerindo que a chaperona seja a
responsável pelo reconhecimento e marcação.
Outro
sistema de grande importância para o reconhecimento de proteínas
malformadas é conhecido como ERAD (Endoplasmic reticulum-associated
protein degradation), responsável pela degradação de proteínas
formadas no retículo endoplasmático, e também realizado com ajuda
de chaperonas.
Após sua
ubiquitinação, as proteínas são mandadas para o citoplasma, onde
encontrarão o proteassoma. A ubiquitinação de uma proteína ocorre
em etapas e envolve 3 enzimas, chamadas de E1 (enzima ativadora da
ubiquitina), E2 (enzima conjugadora) e E3 (ubiquitina ligase).
O processo
consome 1 ATP para a ativação de cada molécula de ubiquitina e
também no desdobramento da proteína realizado pelo proteassoma. A
via da ubiquitina-proteassoma consiste nas seguintes etapas:
1.
Ativação: A enzima ativadora E1 ativa a molécula de
ubiquitina, pela ligação do resíduo C-terminal da ubiquitina a um
resíduo de sulfidrilcisteína da enzima E1. Isso ocorre na presença
de Mg2+ e com o consumo de 1 ATP, liberando AMP e
pirofosfato.
2.
Transferência: A ubiquitina é transferida para a enzima E2,
liberando a enzima E1.
3.
Reconhecimento: A enzima E3 reconhece e se liga à proteína alvo
formando um complexo não-covalente.
4.
Ubiquitinação: O complexo E2-ubiquitina é ligado a E3 de modo
que a ubiquitina seja transferida de E2 para o alvo.
5.
Liberação: a enzima E3 se solta, liberando a enzima E2 e a
proteína ubiquitinada.
6.
Poli-ubiquitinação: As etapas 3, 4 e 5 se repetem várias
vezes, formando uma ou mais cadeias de ubiquitina.
7.
Reconhecimento: a cadeia de ubiquitina é reconhecida pelo
proteassoma. A proteína marcada é desubiquitinada por enzimas
próprias, e desdobrada com consumo de ATP. Após hidrólise, forma
pequenos peptídios, de vida curta, com 7 a 9 aminoácidos. A enzima
E3 (ligase) é a responsável pela especificidade da ubiquitinação,
sendo capaz de reconhecer e marcar um certo conjunto de substratos.
 |
| visão esquemática do processo de ubiquitinação/degradação proteica. |
A
ubiquitinação é reversível; as desubiquinases competem com o
proteassoma, removendo ubiquitinas terminais da cadeia ou a cadeia
inteira. Considera-se que essa competição entre proteólise e
desubiquitinação forneça um certo nível de controle de qualidade
ao sistema, uma vez que a reubiquitinação confirma o reconhecimento
da proteína por degradar.
A
ubiquitinação é reversível, mas não a proteólise. Essa
assimetria permite impor direcionalidade ao sistema, permitindo
assim, por exemplo, o ciclo celular. O mau funcionamento da
ubiquitinação leva a estados patológicos, seja porque não está
degradando proteínas indesejáveis, ou porque está as degradando
muito rapidamente. Enzimas
conhecidas como deubiquitadoras, deubiquitinases (DUBs) ou
isopeptidases podem ainda remover moléculas de ubiquitina ligadas às
proteínas. Como resultado das atividades das DUBs, a ubiquitina
modifica proteínas de modo transitório. Este processo de modificar
dinamicamente as proteínas com ubiquitina cria parâmetros
funcionais reversíveis de um substrato, que permite controlar
numerosos processos celulares. Adicionalmente, algumas DUBs como a
A20, têm dupla função: além de serem capazes de retirar moléculas
de ubiquitina (pelo seu domínio N-terminal) ligadas em resíduo
Lys63, elas podem, alternativamente, ligar outros monômeros (pelo
resíduo C-terminal) em resíduos Lys48 e, dessa forma, controlar não
só o destino da proteína modificada na sua via de sinalização,
como direcioná-la para a degradação pelo proteassoma. É dessa
forma que esse tipo de proteína controla a sinalização
inflamatória induzida por TNF- ou TLR4 modificando os destinos de
TRAF6, RIP1 e IKK e bloqueando a citotoxicidade e apoptose induzida
por ela.
• p53, HPV
e câncer de cerviz: p53 é degradada pela via da ubiquitina
normalmente. Danos no DNA ativam a fosforilação de p53, inibindo a
ubiquitinação. Níveis altos de p53 levam a uma interrupção do
ciclo celular para que o DNA seja reparado. Se o dano for muito
extenso, a célula dispara a apoptose prevenindo a propagação do
erro. O vírus HPV consegue modificar uma proteína E3, a E6-AP, para
que ubiquitine a proteína p53. Dessa forma, ela impede a reparação
celular e leva à formação de tumor no cérvix uterino.
• Reações
imunes: Um fator de transcrição responsável pela reação imune é
ligado a um inibidor enquanto está no citoplasma. Quando a célula é
exposta a bactérias ou substâncias sinalizadoras, a proteína
inibidora é fosforilada, permitindo sua ubiquitinação e a
liberação do fator de transcrição, que é transportado para o
núcleo e ativa alguns genes. A proteólise de proteínas virais
produz peptídios que se apresentam na superfície de células
infectadas, permitindo seu reconhecimento pelos linfócitos T.
• Imunomodulação:
Fragmentos de ubiquitina extracelulares são capazes de diminuir a
resposta imunológica, com efeito comparável à ciclosporina A ou
FK506, drogas amplamente usadas após transplantes de órgãos.
• Imunohistoquímica:
Anticorpos de ubiquitina são usados para identificar acumulação
anormal de proteínas dentro das células, como no mal de Alzheimer e
de Parkinson. Exemplo: o anticorpo PARK2 (Proteína 2 Parkinson, E3-Ubiquitina Proteina Ligase (Parkin)), utilizado na identificação de antígenos ligados à doença de Parkinson.
Um
interessante exemplo do papel do proteassoma foi observado por
Silva-Jardim (2001), em Leishmania chagasi. Em
proteassomas
de Leishmania chagasi
parcialmente
purificados, verificou-se que a função da estrutura no parasita
intacto é necessária para a sua replicação e sobrevivência no
interior da célula do hospedeiro vertebrado.
Sobre a inibição dos proteassomas, há intensa
pesquisa em seu uso clínico. Os inibidores de proteassoma podem
atuar em doenças como diferentes tipos de câncer e várias
aplicações em outras pesquisas. Bortezomib, também conhecido pelo
nome comercial Velcade, é um medicamento aprovado pela FDA e pela
ANVISA e utilizado contra mieloma múltiplo e linfomas de células do
manto. Também é utilizado em pesquisas como inibidor proteassômico.
Existem outros inibidores de proteassomas com potencial terapêutico
e que estão em fase de estudos clínicos, como Disulfiram,
Epigalocatequina-3-galato, Salinosporamida A, Carfilzomib, ONX0912
(Oprozomib), CEP-18770 (Delanzomib) e MLN9708 (Ixazomib).
O
proteassoma faz parte da funcionalidade celular, então inibidores
protessômicos como o MG132 são tóxicos para células e tecidos e
podem causar morte celular em altas concentrações ou após um
tratamento prolongado. É aconselhável titular a concentração
ótima com uma faixa significante. Esta concentração ótima não
depende apenas do tipo celular, mas também dos parâmetros de
cultura como a confluência celular, concentração sérica e
composição do meio.
Também
nos distúrbios associados à obesidade podemos ter participação do
sistema de ubiquitinação proteica. A inabilidade de alguns
neurônios em prevenir o acúmulo de proteínas ubiquitinadas em
inclusões neuronais pode levar à neurodegeneração por meio da
indução de resposta inflamatória. Essa resposta pode ser o fator
gerador, ou ainda ser desencadeada pela expansão de agregados
proteicos anormais no espaço nuclear e citossólico, que não foram
devidamente degradados pelo sistema proteassomal. A importância
desses sistemas proteostáticos nas mais diversas vias
fisiopatológicas começaram a ser caracterizadas na última década,
quando os mecanismos de dano celular foram estreitamente conectados
com a sobrevida e metabolismo dessa célula. Foi nesse contexto que o
trabalho de Ryu, Sinnar et al. (2008) caracterizarou um fenótipo de
normofagia, hiperleptinemia e obesidade na idade adulta de animais
experimentais que sofreram a deleção do gene Ubb, o qual é
encarregado de codificar as ubiquitinas. O knockout
gênico
provocou perda de 30% de neurônios predominantemente do núcleo
arqueado, colocalizando proteínas ubiquitinas e neurônios
produtores de NPY, AgRP e POMC, associadas a gliose persistente além
de distúrbios no sono.

Comentários
Postar um comentário